

Article du magazine SEMPER - édition mars 2021 – www.dsb.lu
Rubrique sous la responsabilité du Dr Manon Gantenbein, PhD, Responsable du CIEC, LIH
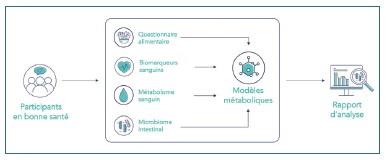
Nutriomix est une étude initiée par le département de Bioinformatique (BioCore) du centre de recherche interdisciplinaire ‘Luxembourg Center for Systems Biomedicine’ (LCSB) rattaché à l’Université du Luxembourg. Cette étude financée dans le cadre d’un programme “Proof Of Concept” a pour objectif d’étudier la faisabilité du développement d’une offre de suivi nutritionnel par l’analyse de biomarqueurs sanguins, du métabolome et du métagénome issu de la flore bactérienne intestinale et la réalisation de recommandations nutritionnelles suivant le profil d’habitudes alimentaires via la modélisation et de la simulation métabolique à partir de ces données.
A ce titre, il s’agit de la première étude au Luxembourg basée sur une approche de “phénotypage profond” en nutrition et elle pourrait constituer une pierre angulaire pour de futures recherches dans le domaine. A terme, cette offre pourrait être intégrée à une application mobile permettant à tout un chacun (i) de planifier des repas équilibrés et sains, adaptés à son profil métabolique et (ii) de suivre les recommandations nutritionnelles qui leur sont proposées par les professionnels de santé.
Nutriomix, qui a été autorisée par le Ministère de la Santé en février 2020, est une étude pilote à double titre. D’une part, elle constitue la première étude observationnelle sur volontaires sains du département de Bioin¬formatique du LCSB. D’autre part, son caractère pilote lui est conférée par le fait qu’elle est centrée sur les aspects logistiques et de faisabilité du traitement des données pour envisager le cas échéant une étude consécutive à plus large échelle. Cependant, et bien que considérée comme pilote, cette étude réunit 7 entités présentes sur les 4 pays que constituent le Luxembourg et ses pays voisins limitrophes.
Nos sociétés modernes sont confrontées à un nombre croissant de maladies non transmissibles (MNT) et en particulier de maladies chroniques, ce qui représente un fardeau humain et économique considérable. Elles sont les principales causes de décès, de maladie et d’invalidité dans la région européenne de l’OMS. Et pourtant, certaines d’entre elles seraient évitables ou retardées en adoptant des habitudes alimentaires vertueuses.
Avec les récents progrès scientifiques sur le plan de la connaissance du métabolisme, il est apparu clairement que la manière dont on réagit à la nourriture varie considérablement d’un individu à l’autre. Une personnalisation pourrait alors être envisageable dans le but d’ajuster les apports nutritionnels suivant le profil métabolique de chacun.
Cependant, pour qu’une démarche d’intervention alimentaire soit envisagée, il convient d’évaluer les apports au plus près de l’alimentation en condition de vie réelle et d’utiliser des marqueurs appropriés permettant de fournir des indications sur la façon dont notre corps digère les aliments et produit les métabolites. Certains de ces marqueurs sont «statiques», comme le génome individuel (c’est-à-dire la constitution génétique héréditaire d’un individu), qui peut influencer l’efficacité de la digestion de certains nutriments ou la prédisposition individuelle à la prise de poids.
D’autres facteurs sont par contre dynamiques et changent avec le temps, comme l’environnement, le niveau d’exercice ou la flore bactérienne intestinale. Cette dernière joue un rôle clé dans la digestion, en aidant à dégrader certains nutriments que notre métabolisme humain n’est pas capable de faire seul. En effet, la composition de cette communauté bactérienne, appelée microbiote intestinal, a été associée aux habitudes alimentaires et à plusieurs maladies.
Ainsi, en mesurant les différents paramètres précités, il serait possible de formuler une stratégie nutritionnelle mais aussi d’obtenir des infor¬mations au niveau moléculaire sur l’efficacité d’une intervention alimentaire, tant pour des individus bien portant animés par une démarche de maintien de leur bonne santé, que pour des individus porteurs de pathologies associées ou impactées par l’alimentation.
Le concept de régime alimentaire “unique” semble pouvoir laisser la voie à des approches personnalisées plus en phase avec les besoins des individus et des professionnels de santé souhaitant explorer ces options. En ce qui concerne Nutriomix, cette étude pilote se focalise sur les voies métaboliques et le microbiote intestinal et dont les résultats serviront, entre autres choses, à structurer une offre qui sera proposée par la startup luxembourgeoise Nium, une spin-off du LCSB.
Les approches actuelles en matière de nutrition personnalisée sont basées sur des techniques d’analyse de données (par exemple, méthodes statistiques, apprentissage automatique) et sur les connaissances des professionnels de la santé/nutrition. Toutefois, ces approches ne tiennent pas compte des connaissances accumulées sur les voies métaboliques présentes dans l’organisme humain ou les bactéries intestinales. L’équipe du LCSB, et l’équipe Nutriomix en particulier, ont largement contribué (souvent en tant qu’auteurs principaux) au développement de ces connaissances sous la forme de reconstructions métaboliques à l’échelle du génome.
Il est important de noter qu’il est possible d’utiliser ces modèles pour intégrer des données diététiques, microbiologiques et métabolomiques. La composition microbiotique individualisée de l’intestin est dérivée des données de la métagénomique (obtenues par séquençage de l’ARN ribosomal 16s ou de l’ADN entier) des échantillons de selles. Les informations alimentaires sont établies en convertissant le contenu nutritionnel des aliments en quantités de métabolites. Cette méthodologie permet d’évaluer l’impact des habitudes alimentaires actuelles sur le système et de comprendre les variations individuelles en réponse à une composition alimentaire spécifique.
Ainsi, pour évaluer la variabilité individuelle en réponse au régime alimentaire par la modélisation métabolique et l’intégration des données métabolomiques (métabolites du sang) et du microbiome intestinal (analyse génétique des bactéries intestinales), il a été décidé de mettre en oeuvre une étude observationnelle transversale non interventionnelle avec un maximum de 20 participants volontaires sains qui ont été invités à fournir un échantillon sanguin et de leurs selles et à compléter un questionnaire sur leurs habitudes alimentaires.
Pour la mise oeuvre du recrutement des participants, la gestion de la visite avec le médecin de l’étude et la récolte des données, le suivi général des participants, le Centre d’investigation et d’Épidémiologique Clinique (CIEC) du Luxembourg Institute of Health (LIH), dirigé par le Dr Manon Gantenbein, a été mandaté pour sa qualité et son expertise ainsi que pour la compétence de ses équipes dans l’accompagnement des participants au remplissage du questionnaire. En effet, le questionnaire choisi est celui qui a été utilisé notamment pour l’étude ORISCAV au Luxembourg.
Les échantillons biologiques ont, quant à eux, été récoltés par le Laboratoire Ketterthill qui s’est chargé de réaliser les analyses des biomarqueurs sanguins, et de transmettre les aliquots du métabolome et du microbiome pour analyse auprès de laboratoires tiers.
Au terme de l’analyse des données, les participants recevront un rapport d’analyse. De son côté, la Startup Nium effectuera la modélisation et l’intégration les données pseudonymisées afin d’observer les voies mé¬taboliques associées à la variabilité individuelle et de valider la faisabilité technique et logistique d’une possible offre adaptée au besoin et au contexte d’un suivi nutritionnel.
NIUMLa startup NIUM est une jeune spin-off de l’Université du Luxembourg opérant dans le domaine de la nutrition personnalisée. Elle a été co-fondée par le Dr Alberto Noronha, Bioinformaticien et le Dr Adam Selamnia, Biochimiste et entrepreneur en e-santé, agissant respectivement en qualité de Gérant et Business Advisor. NIUM associe des technologies de modélisation métabolique (Metabolic Digital Twin) et algorithmiques de recommandations nutritionnelles dans le but de permettre aux individus de faire les meilleurs choix alimentaires pour une vie plus saine. Elle lancera dans les prochaines semaines son application mobile Nutrida au Luxembourg. Cette application permettra à ses utilisateurs de planifier leur repas à base de recettes, faciles à cuisiner et utilisant des produits de saison, parmi lesquelles certaines sont proposées par des chefs ou des cuisiniers reconnus. Dans un second temps, il sera possible de commander des tests métaboliques auprès des laboratoires au Luxembourg pour monitorer sa santé, le cas échéant avec son professionnel de santé, et de commander les ingrédients sur les sites d’e-commerce des distributeurs directement depuis l’application. La startup a participé au programme d’accélération de startups au Luxembourg Fit4Start et au programme européen EIT Food Acceleration Network. Elle est actuellement accompagnée par EY dans le cadre de son programme EYNovation.
|
Les données récoltées pour chaque participant sont en cours d’analyse:
• 50 biomarqueurs sanguins et sériques ont été testés,
• 180 métabolites du métabolome constitués d’acides aminés, d’amines bioactives et d’acides gras complexes (40 acylcarnitines, 42 amino acids/biogenic amines, 91 phospholipids, 15 sphingolipids),
• plus de 800 souches bactériennes seront analysées pour la création des profils bactériens et l’étude des flux métaboliques suivant leur pré-pondérance dans l’écosystème reconstitué.
Les 180 métabolites testés constituent des molécules qui ont un intérêt dans le domaine nutritionnel. De même, les 800 souches bactériennes ana¬lysées dans l’étude comptent parmi celles qui sont fréquemment retrou¬vées dans les selles, et surtout pour lesquelles il a été possible de recréer l’ensemble des voies métaboliques à partir de la connaissance de leur gé¬nome.
Les résultats de cette étude sont attendus avant la fin du semestre en cours. Le cas échéant, ils permettront de mieux appréhender le degré de données utiles pour accompagner les programmes de suivis nutritionnels et les flux à envisager en prenant en compte les questions de respect de la vie privée et de la sécurité informatique.
A ce titre, une réflexion sera menée sur la constitution de panel de tests d’intérêt en fonction de profils alimentaires, par exemple afin de vérifier l’impact positif attendu des recommandations de recettes. Enfin, Il sera nécessaire de prendre en compte la réalité et la faisabilité économique, en particulier la dépense associée à ces tests, afin de proposer à terme un service inclusif et accessible aux populations souhaitant intégrer une approche préventive de leur santé.
Le LCSB rattaché à l’Université du Luxembourg est le sponsor de l’étude et Pr. Reinhard Schneider, Directeur de l’Unité Bioinformatics Core, en est l’Investigateur Principal. Les Drs Alberto Noronha et Adam Selamnia, désormais opérant au sein de la spin-off Nium (voir l’encadré ci-dessus), ont été les chefs de projets successifs et leur startup est chargée de la gestion opérationnelle et de l’analyse des résultats de l’étude Nutriomix. La gestion du recrutement et du suivi des participants a été confiée au CIEC du LIH. Le questionnaire sur les habitudes alimentaires a été fourni par le Département des Sciences de la Santé publique de l’Université de Liège (Belgique) sous la direction du Pr Michèle Guillaume. Enfin, les analyses des biomarqueurs sanguins et du métabolome ont été respectivement confiées au Dr Sylvie Coito, Médecin Biologiste du Laboratoire Ketterthill et au Max Planck Institute (Marburg, Allemagne); et celles du microbiome au Laboratoire Cerba (Saint-Ouen l’Aumone,
for articles/videos/studies
The articles can be sorted by therapeutic area or disease, but may also deal with more general topics not specifically related to a disease. These articles can be sorted as "other".